Apuntes
Diseñan en el CICESE péptido antibacteriano con novedosas herramientas computacionales
Logran el primer diseño computacional de un péptido antibacteriano con un paradigma de diseño novedoso, desarrollado en el CICESE
 Este diseño por computadora, hecho en el CICESE, se probó en la Universidad de Pensilvania.
Este diseño por computadora, hecho en el CICESE, se probó en la Universidad de Pensilvania.
ENSENADA.- En el Laboratorio de Biología Computacional del Centro de Investigación Científica y de Educación Superior de Ensenada, Baja California (CICESE), se logró el primer diseño computacional de un péptido antibacteriano con un paradigma de diseño novedoso, desarrollado en este centro de investigación.
Aunque los productos naturales siguen siendo una fuente valiosa de nuevos fármacos, su desarrollo y optimización mediante síntesis química suelen requerir largos procesos experimentales.
La rápida evolución de las enfermedades, la aparición de nuevas pandemias y el aumento de la resistencia a los antibióticos, subrayaron la necesidad urgente de diseñar terapias eficaces en menor tiempo.
Los enfoques computacionales permiten la generación de metodologías, técnicas y herramientas que facilitan y aceleran el modelado y diseño molecular. Así, observamos representaciones tridimensionales de moléculas, construcción de redes de interacción entre estas moléculas y sus blancos. Esto equipado con tecnología de inteligencia artificial para encontrar nuevos fármacos.
El diseño computacional de proteínas y péptidos está contribuyendo a transformar la ingeniería de macromoléculas con estructuras y funciones precisas. Sin embargo, los métodos actuales se basan predominantemente en modelos generativos, es decir, en inteligencia artificial que crea nuevos datos a partir de patrones aprendidos en conjuntos de datos existentes, para generar nuevo contenido, pero que exigen una gran demanda en recursos computacionales, que son costosos para entrenar y modificar.
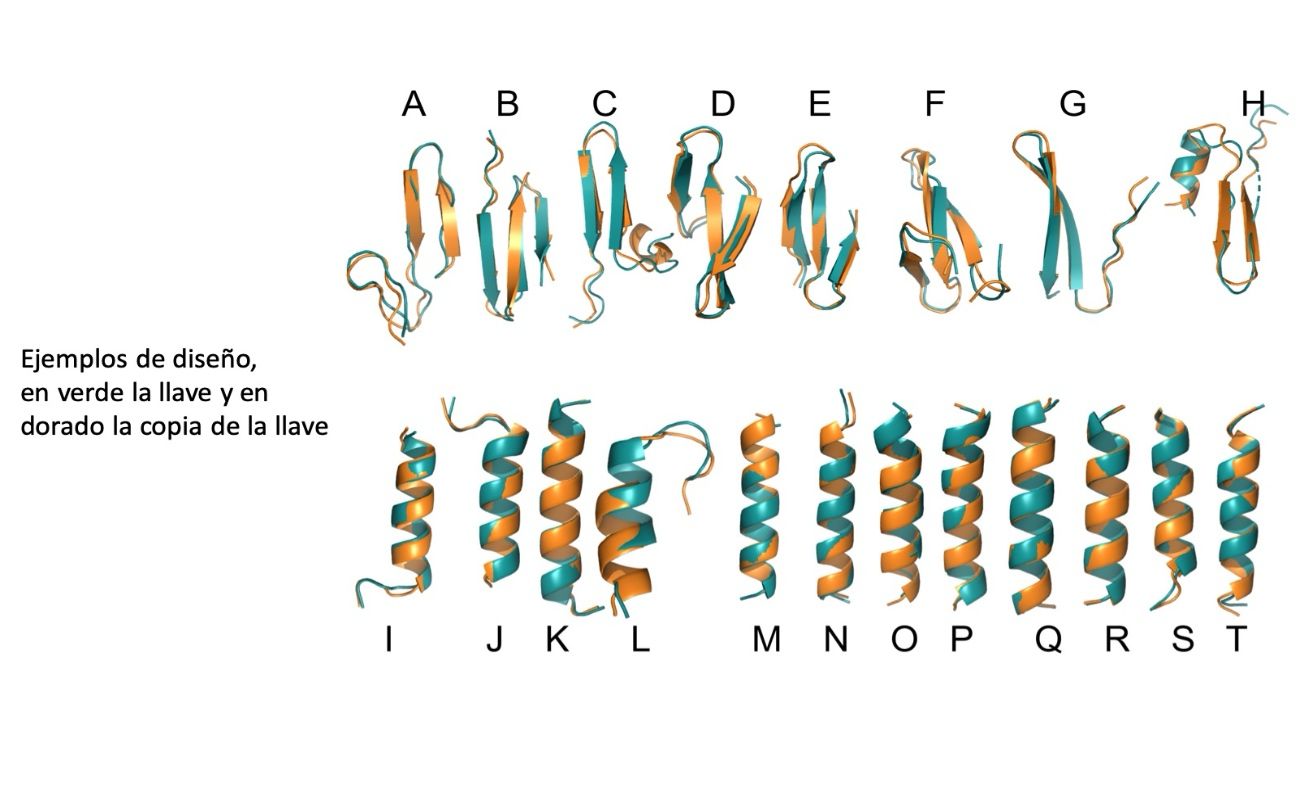
Cual cerrajero con su máquina copia llaves, KCM–CICESE copia un péptido antibacteriano
Frente a esto, en el CICESE se creó un paradigma de diseño denominado, máquina de copia de llaves (Key Cutting Machine –KCM), una plataforma basada en la optimización, o sea, enfocada en hallar la mejor solución posible a un problema, que aprovecha iterativamente la predicción de la estructura de la secuencia diseñada (la copia de llave), para que esta coincida con la geometría de la estructura que se quiere copiar (la llave).
“Si tienes una idea del tipo de llave que necesitas, nuestra máquina puede hacer una copia. En nuestro caso, la llave es un péptido antibacteriano y la copia es otro péptido antibacteriano, pero con una secuencia de aminoácidos totalmente diferente al de la llave original”, explica el doctor Carlos Alberto Brizuela Rodríguez, investigador del Departamento de Ciencias de la Computación del CICESE.
A diferencia de los modelos generativos que requieren una gran cantidad de datos de entrenamiento y consecuentemente varios procesadores gráficos (GPUs), KCM requiere una sola unidad de GPU y, lo más relevante, permite la incorporación directa en el diseño de cualquier requisito definido por el usuario, a través de la función objetivo. Así, se evitan los altos costos de reentrenamiento típicos de los modelos generativos y facilita una evaluación directa de las propiedades medibles.
Mediante el empleo de un algoritmo de estimación de distribución, KCM optimiza las secuencias con base en criterios geométricos, fisicoquímicos y energéticos, favoreciendo un diseño preciso. Evalúa la calidad de cada candidato y utiliza esta evaluación para guiar las mejoras posteriores.
En el caso del desarrollo en el CICESE, el diseño arrojó un candidato: un péptido, con potente actividad contra múltiples cepas bacterianas y eficacia en un modelo de infección causada por patógenos en ratones.
Para probar el péptido antibacteriano diseñado por computadora en el CICESE, se trabajó colaborativamente con un grupo de investigación de la Universidad de Pensilvania donde el péptido antibacteriano fue probado in vitro y en pruebas preclínicas obteniendo un éxito comparable a los antibióticos comerciales.
Este resultado fue publicado recientemente en la revista Nature Machine Intelligence bajo la firma de Yan C. Leyva, Marcelo D.T. Torres, Carlos A. Oliva, César de la Fuente Nuñez y Carlos A. Brizuela, el primero, el tercero y último autores, respectivamente, son egresado de la maestría, estudiante de doctorado, e investigador titular en el Departamento de Ciencias de Computación del CICESE.
El artículo “Tailored structured peptide design with a key-cutting machine approach” es de acceso libre y puede consultarse en Nature Machine Intelligence (https://www.nature.com/articles/s42256-025-01119-2).
Al combinar accesibilidad y precisión nanométrica, el KCM se posiciona como un potente modelo para diseñar péptidos de novo, sin la necesidad de hardware de alta gama, con aplicaciones en medicina, ciencia de materiales, biología sintética, y accesible para hacer investigación en laboratorios pequeños.
Las proteínas de novo son proteínas diseñadas desde cero en un laboratorio, con secuencias de aminoácidos completamente nuevas, no encontradas en la naturaleza, para cumplir funciones específicas y aplicables en la obtención de fármacos y vacunas, entre otras.
Por lo tanto, concluye el artículo científico, el KCM representa una herramienta robusta para el diseño de proteínas y péptidos de novo, ofreciendo un paradigma flexible para replicar y extender las relaciones estructura–función de las plantillas existentes.
TAGS
TE PUEDE INTERESAR












